我目前数据分析的主力编程语言是python,使用jupyter notebook而非Rstudio作为我的编程环境。当然R语言也不能丢,好在python提供了rpy2使得我能在python调用R。
第一步是,安装rpy2
pip intall rpy2
接着,在jupyter notebook中导入相关的环境和一些简单的配置
import rpy2.rinterface_lib.callbacks
from rpy2.robjects import pandas2ri
# Ignore R warning messages
#Note: this can be commented out to get more verbose R output
rpy2.rinterface_lib.callbacks.logger.setLevel(logging.ERROR)
# 这一步是将R输出的data.frame转成Pandas的DataFrame
pandas2ri.activate()
当然,下面才是非常关键的一步!
%load_ext rpy2.ipython
他有如下的作用
- 加载 RPy2 扩展: 这使得 R 语言的功能可以在 Python 环境中使用,提供了一种混合这两种语言编程的能力。
- 提供了额外的魔术命令: 例如
%R用于在单个代码单元中执行 R 代码,%%R用于在整个代码单元中执行 R 代码,以及%Rpush和%Rpull用于在 Python 和 R 之间传输变量。 - 方便的数据转换: 当使用 RPy2 时,可以轻松地将 Python 中的数据结构(如 Pandas DataFrame)转换为 R 中的数据结构(如 R 的 data.frame),反之亦然。
于是乎,我们就能加载R库了
%%R
# Load libraries from correct lib Paths for my environment - ignore this!
# .libPaths(.libPaths()[c(3,2,1)])
# Load all the R libraries we will be using in the notebook
library(clusterProfiler)
library(ggplot2)
举个简单的例子,使用clusterProfiler进行数据分析
%%R
library(org.Hs.eg.db)
data(geneList, package="DOSE")
gene <- names(geneList)[abs(geneList) > 2]
ego <- enrichGO(gene = gene,
universe = names(geneList),
OrgDb = org.Hs.eg.db,
ont = "CC",
pAdjustMethod = "BH",
pvalueCutoff = 0.01,
qvalueCutoff = 0.05,
readable = TRUE)
dotplot(ego)
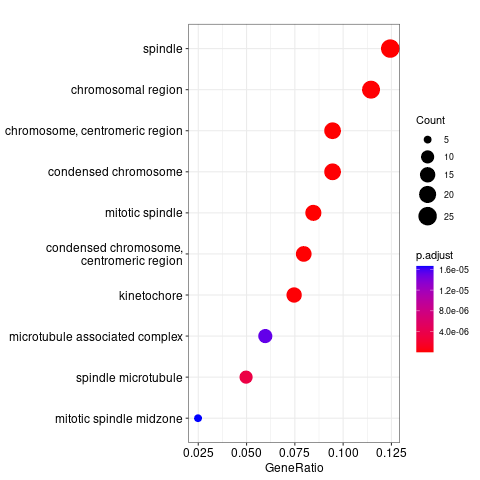
需要注意的是,复杂的自定义的数据结构,是不可能从R中转换到python的,例如ego,就是clusterProfiler自定义的数据结构,但是我们可以将其转成数据框(data.frame)这类R的基本数据结构。
%%R -o enrich_result
enrich_result = as.data.frame(ego)
这里我们在%%R后添加了-o enrich_result表明输出结果的enrich_result是需要导出的python的对象。
类似,假设你有一组python的gene_list,可以通过-i gene_list导入到R中。
%%R -i gene_list
print(gene_list)
