太长不看版: 文献报道XIST和RPS4Y1是区分性别的两个高可信度的标记基因,因此你没有必要去用其他性染色体上的基因去确定数据集的性别。
不仅仅是在使用公共的单细胞转录组数据,其实早在公共芯片数据或者RNA-seq数据挖掘中,就有人在考虑一个问题,这个数据的元信息作者会不会搞错了呢?
以性别为例,我们很容易想到表达Y染色体上基因数据肯定是男性,但是我们也知道基因也不是任何时刻都表达,所以如果一个Y染色体上的基因不表达,ta未必是女性。因此我们需要一个比较可靠的标记基因,来确保对性别的区别是正确的。
我最初的想法,也是对Y染色体的基因逐个看表达,但是转念想到,在我这个数据集中有用的标记未必适用于其他数据集呀。因此通过一波检索,我找到了一篇文献,里面给出了两个关键基因,XIST和RPS4Y1。

接着我用Seurat提供的一个公共数据集进行测试,这个数据包括了不同技术处理的PBMC数据,预处理的代码如下。
library(Seurat)
library(harmony)
data("pbmcsca")
library(dplyr)
pbmc <- pbmcsca%>%
Seurat::NormalizeData(verbose = FALSE) %>%
FindVariableFeatures(selection.method = "vst", nfeatures = 2000) %>%
ScaleData(verbose = FALSE) %>%
RunPCA(pc.genes = pbmc@var.genes, npcs = 20, verbose = FALSE)
pbmc <- RunHarmony(pbmc, c("Experiment", "Method"))
pbmc <- RunUMAP(pbmc, reduction = "harmony", dims = 1:20)
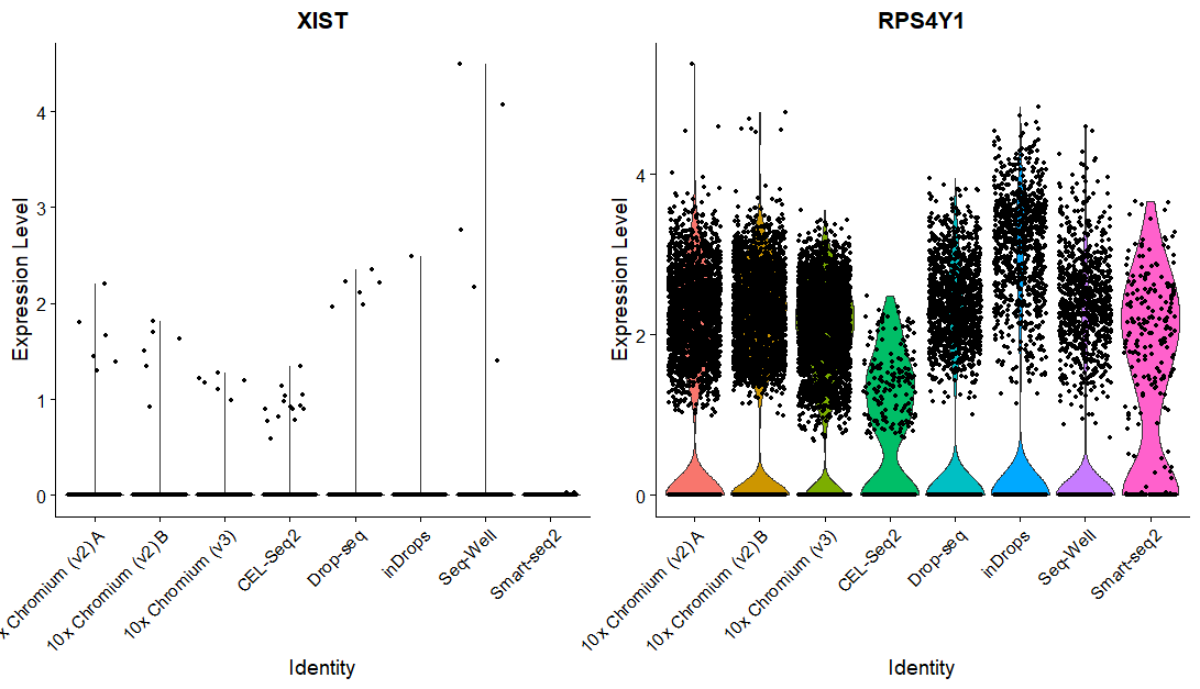
最终我们获得了使用harmony去除批次效应后的数据集,接着我们用小提琴图分来源对XIST和RPS4Y1进行可视化
VlnPlot(pbmc, c("XIST","RPS4Y1"), group.by = "Method")
结果如下

你会很奇怪为什么CEL-Seq2, Drop-Seq, InDrops, Seq-Well,Smart-seq2什么同时表达这两个基因呢?
很简单,因为这几种方法同时还包括两种实验,pbmc1和pbmc2

当我们筛选所有的pbmc1实验进行展示
pbmc_sub <- subset(pbmc, Experiment == "pbmc1")
VlnPlot(pbmc_sub, c("XIST","RPS4Y1"), group.by = "Method")
你会发现这两个是完美的互斥关系

如果你筛选出pbmc2进行展示
pbmc_sub <- subset(pbmc, Experiment == "pbmc2")
VlnPlot(pbmc_sub, c("XIST","RPS4Y1"), group.by = "Method")
同样的,你得到一个完美的互斥结果

小结: XIST和RPS4Y1是区分性别的两个高可信度的标记基因,如果以后使用人的公共数据集的时候,可以用这个两个基因确定性别。
参考资料: