本文主要介绍如何使用JCVI的synteny子命令基于已有的共线性分析结果,展现局部的共线性。
需要准备的三个输入文件
- 记录物种内或者物种间的共线性基因对
- 记录基因坐标的bed文件
- 布局文件
第一步,基于已有的共线性分析结果(WGDI, MCscan, MCscanX等软件的分析结果),整理出你需要展示的区间的基因对。注意分隔符是制表符,我们保存为blocks.txt
AL1G16390 AT1G06380
AL1G16400 AT1G06390
AL1G16410 AT1G06400
AL1G16420 AT1G06410
AL1G16430 AT1G06420
AL1G16440 AT1G06430
AL1G16450 AT1G06440
AL1G16460 AT1G06450
AL1G16470 AT1G06460
AL1G16480 AT1G06470
AL1G16490 AT1G06475
AL1G16510 AT1G06490
AL1G16520 AT1G06500
AL1G16530 AT1G06515
AL1G16540 AT1G06510
AL1G16550 AT1G06520
AL1G16560 AT1G06530
AL1G16570 AT1G06540
AL1G16580 AT1G06550
AL1G16590 AT1G06560
AL1G16600 AT1G06570
AL1G16610 AT1G06580
第二步,整理记录基因坐标的bed文件。bed要求是6列,记录基因的坐标和朝向,第五列填0即可。 我们命名为genes.bed
1 3631 5899 AT1G01010 0 +
1 6788 9130 AT1G01020 0 -
...
scaffold_9 1792941 1795545 AL9U12210 0 -
scaffold_9 1796870 1801565 AL9U12220 0 -
scaffold_9 1810180 1812874 AL9U12230 0 +
scaffold_9 1836100 1837535 AL9U12240 0 -
两个要求:
- 第4列的基因名必须对应共线性对的基因
- 文件里必须包含你需要展示物种的所有基因(至少是共线性区块的基因)
第三步,提供布局文件, 命名为layout.csv
# x, y, rotation, ha, va, color, ratio, label
0.5, 0.4, 0, center,top, , 1, A.lyrata Chr1
0.5, 0.3, 0, center, top, , 1, A.thaliana Chr1
# edges
e, 0, 1
该文件分为两个部分:上半部分是track在图中的相对位置(x,y)和旋转角度(rotation),以及label的对齐方式, ha( left, center, right) va( top, button) 和颜色(color)
下半部分是不同track的共线性关系,e,0,1表示第一个和第二个track有关联。
最后运行程序
python -m jcvi.graphics.synteny blocks.txt genes.bed layout.csv
输出结果为一个pdf,如下所示
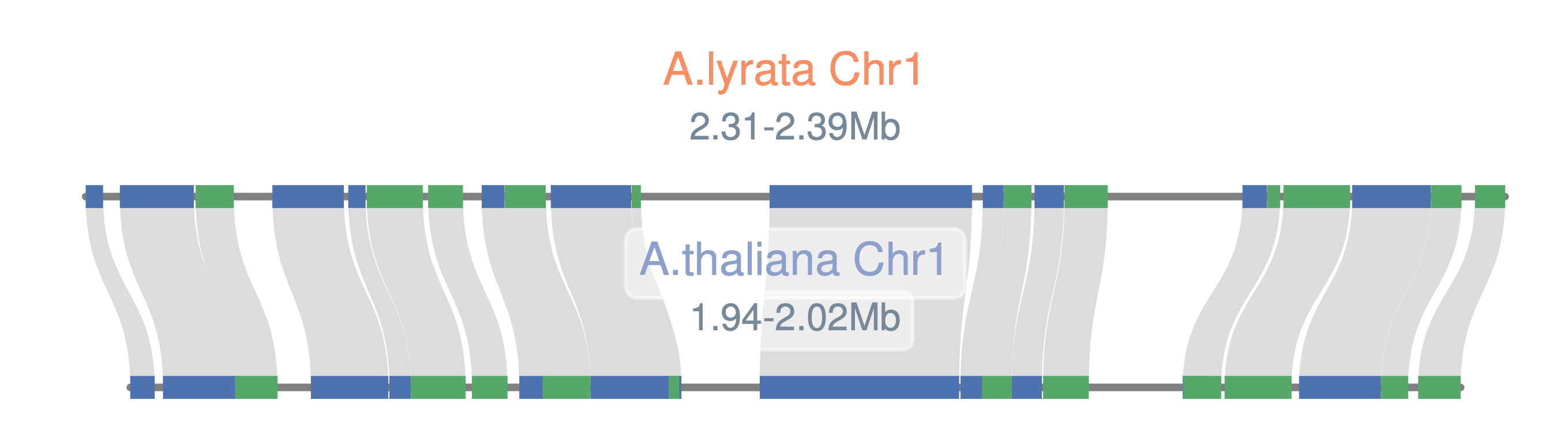
因为我们的共线性基因里面是A.lyrata是第一列,所以画图的时候也是A.lyrata在第一行。
案例仅展示了2条序列之间的共线性,实际上jcvi.graphics.synteny是可以展示多条序列的结果,比如说jcvi案例展示graph, peach, cacao三者之间的共线性,提供的两两之间的共线性如下
GSVIVT01012261001 . .
GSVIVT01012259001 . .
GSVIVT01012258001 . .
GSVIVT01012257001 . .
GSVIVT01012255001 Prupe.1G290900.1 Thecc1EG011472t1
GSVIVT01012253001 Prupe.1G290800.2 Thecc1EG011473t1
GSVIVT01012252001 Prupe.1G290700.1 Thecc1EG011474t1
GSVIVT01012250001 Prupe.1G290600.1 Thecc1EG011475t1
GSVIVT01012249001 Prupe.1G290500.1 Thecc1EG011478t1
GSVIVT01012248001 Prupe.1G290400.1 Thecc1EG011482t1
通过调整布局(注意布局文件里面的坐标x,y, 以及edge)
# x, y, rotation, ha, va, color, ratio, label
0.5, 0.6, 0, center, top, , 1, grape Chr1
0.3, 0.4, 0, center, bottom, , .5, peach scaffold_1
0.7, 0.4, 0, center, bottom, , .5, cacao scaffold_2
# edges
e, 0, 1
e, 0, 2
就能输出如下效果的共线性
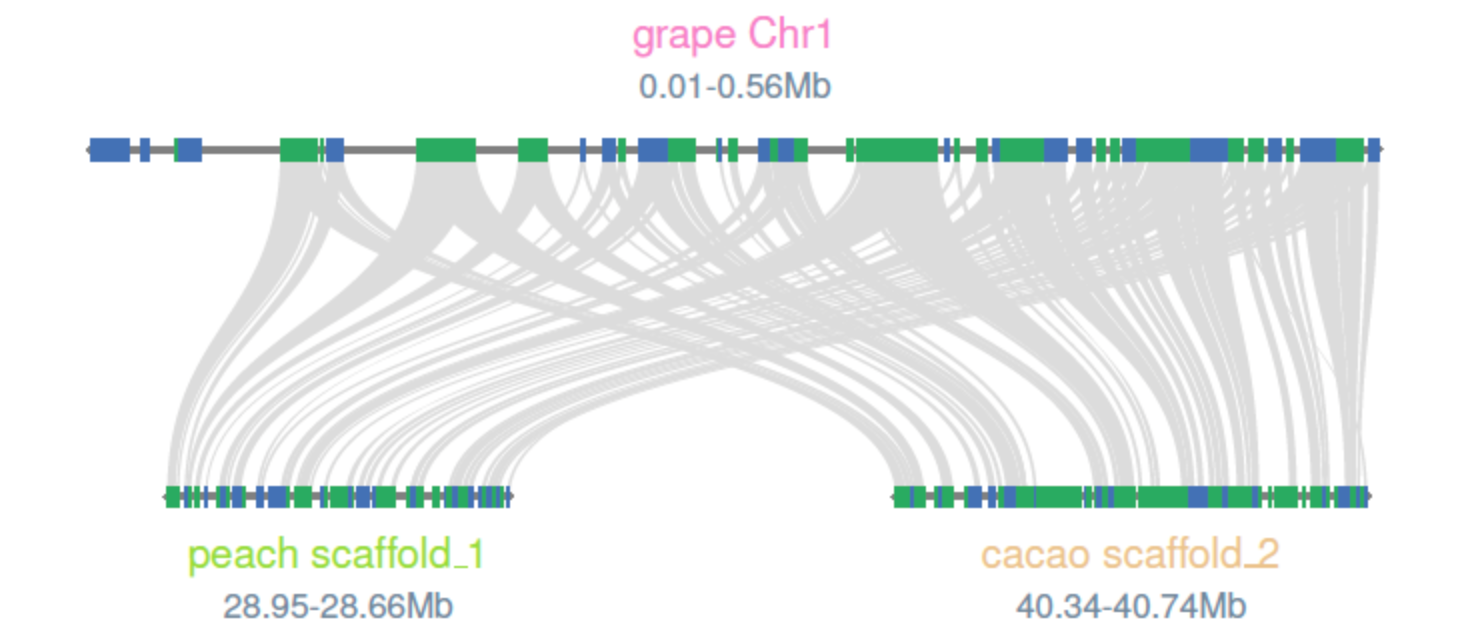
参考资料
https://github.com/tanghaibao/jcvi/wiki/MCscan-(Python-version)
