一句话评价:重复序列注释用EDTA就完事了。
简介
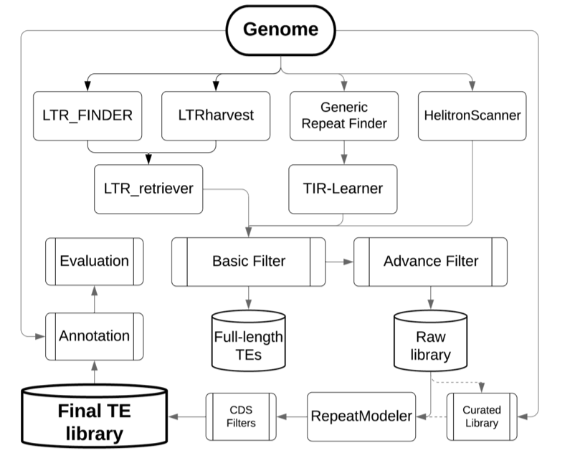
EDTA, 全称是 Extensive de-novo TE Annotator, 一个综合性的流程工具,它整合了目前LTR预测工具结果,TIR预测工具结果,MITE预测工具结果,Helitrons预测工具结果, 从而构建出一高可信,非冗余的TE数据库,用做基因组的注释。流程图如下

安装
如果没有管理员权限,可以用conda进行安装。如果有管理员权限,可以尝试用docker或者singularity进行安装。
PS: 如果之前用过EDTA的话,可以更新一下版本,因为从1.7.0开始,EDTA把intact TE和homology based注释结合在一起,最终产生了很高质量的gff,合并了所有注释;1.7.1版把包含基因的intact也去掉了,进一步过滤。(来自于作者的建议)
conda
使用conda的安装方法如下
conda create -n EDTA
conda activate EDTA
python2 -m pip install --user numpy==1.14.3 biopython==1.74 pp
conda config --env --add channels anaconda --add channels conda-forge --add channels biocore --add channels bioconda --add channels cyclus
conda install -n EDTA -y cd-hit repeatmodeler muscle mdust repeatmasker=4.0.9_p2 blast-legacy java-jdk perl perl-text-soundex multiprocess regex tensorflow=1.14.0 keras=2.2.4 scikit-learn=0.19.0 biopython pandas glob2 python=3.6 trf
git clone https://github.com/oushujun/EDTA
./EDTA/EDTA.pl
需要注意的一点是,bioconda建议添加国内镜像站点, 否则可能会下载失败。
singularity
方法1: 使用EDTA上提供的docker镜像,以singularity进行安装
singularity build edta.sif docker://kapeel/edta
在使用时,有一点需要注意,需要用-B将外部的RepeatMasker的Libraries绑定的Libraries,否则可能会在检查依赖这一步失败。
这里,我参考的是LoReAN的方法
cd ~ # 切换到家目录
mkdir -p LoReAn && cd LoReAn
wget https://github.com/lfaino/LoReAn/raw/noIPRS/third_party/software/RepeatMasker.Libraries.tar.gz && tar -zxvf RepeatMasker.Libraries.tar.gz
singularity exec \
-B /home/xzg/LoReAn/Libraries/:/opt/conda/share/RepeatMasker/Libraries/ \
/home/xzg/edta.sif /EDTA/EDTA.pl -h
上面的/home/xzg/是我的家目录,需要根据实际情况进行选择
方法2: 使用 @wangshun1121 构建的docker镜像, 他解决了需要-B进行挂载的问题。
singularity build edta.sif docker://registry.cn-hangzhou.aliyuncs.com/wangshun1121/edta
实战
我们以拟南芥的第一条染色体为例,进行介绍
# singularity
singularity exec ~/LoReAn/edta.sif /EDTA/EDTA.pl \
-genome chr1.fa -species others -step all -t 20
# conda
# 我的EDTA在我的家目录下
~/EDTA/EDTA.pl -genome chr1.fa -species others -step all -t 20
这里的参数比较简单,
-genome: 输入的基因组序列-species: 物种名,Rice, Maize和others三个可选-step: 运行步骤,all|filter|final|anno, 根据具体情况选择-t: 线程数,默认是4
此外还有几个参数可以关注下
-
-cds: 提供已有的CDS序列(不能包括内含子和UTR),用于过滤.这个值也比较重要,建议提供下,否则会降低busco值(来自于作者的推荐) -
-sensitive: 是否用RepeatModeler分析剩下的TE,默认是0,也就是不要。RepeatModeler运行时间比较久,量力而信。 -
-anno: 是否在构建TE文库后进行全基因组预测,默认是0. -
-evalues 1: 默认是0,需要同时设置-anno 1才能使用。建议加上,它能够查看注释质量,是非常不错的功能哦(来自于作者的推荐)
运行结束之后,会在当前目录下留下运行时的中间文件,保证你程序中断之后,能够断点续跑 -
xxx.EDTA.raw
-
xxx.EDTA.combine
-
xxx.EDTA.final
以及你关注的xxx.EDTA.TElib.fa, 这就是最终的TE文库。
需要注意的是,在实际运行的时候,你不能单条染色体的运行,这不是程序设计的目的,我们这里用一条染色体仅仅是为了演示,测试程序能否顺利运行。
而在实际项目中,一定要用所有的染色体或者scaffold
可能问题
我在使用EDTA时,就遇到了两个问题。一个是singularity的EDTA直接使用时无法通过依赖检测,解决方法已经在安装部分提过,这里不在赘述。
另一个问题我在"Identify TIR candidates from scratch"这一步出现下面的报错
what(): terminate called after throwing an instance of 'Resource temporarily unavailable std::system_error'
what(): Resource temporarily unavailable
terminate called after throwing an instance of 'std::system_error'
我对这个报错进行了分析,找到了对应代码,即sh $TIR_Learner -g $genome -s $species -t $threads -l $maxint. 用实际内容替换变量后,即下面这行代码
sh /EDTA/bin/TIR-Learner2.4/TIR-Learner2.4.sh -g chr.fa -s others -t 20 -l 5000
更具体一点,可以将问题定位到脚本的Module 3, Step 3: Get dataset
genomeFile=/data/xzg_data/1800_assembly/annotation/repeatAnnotation/chr.fa #基因组文件的实际路径
genomeName=TIR-Learner
tir_path=/EDTA/bin/TIR-Learner2.4 # TIR-Learner2.4的路径
t=1
dir=`pwd`
export OMP_NUM_THREADS=1
python3 $tir_path/Module3_New/getDataset.py -g $genomeFile -name $genomeName -p $tir_path -t $t -d $dir"/Module3_New"
将线程数设置为1后,该代码顺利跑通。进一步,我定位getDataset.py的出问题的地方实际是predict函数。当然接着执行后续的代码,发现改动这一参数并不影响下面代码的运行。
echo "Module 3, Step 4: Check TIR/TSD"
python3 $path/Module3_New/CheckTIRTSD_M3.py -name $genomeName -p $path -t $t -d $dir"/Module3_New"
echo "Module 3, Step 5: Write to Gff"
python3 $path/Module3/WriteToGff_M3.py -name $genomeName -p $path -t $t -d $dir"/Module3_New"
我发现predict函数涉及到了Python的多进程调用,最终在偶然间找到问题真正所在,即Linux系统对用户的资源限制,可以通过ulimit -a查看。
最终我通过设置ulimit -u 9000,提高允许运行的总程序数,将问题解决。
参考资料
- EDTA官方文档
- RMblast的问题
- 一个关于fork资源不够的解决过程
- Ou, S., Su, W., Liao, Y., Chougule, K., Agda, J.R.A., Hellinga, A.J., Lugo, C.S.B., Elliott, T.A., Ware, D., Peterson, T., et al. (2019). Benchmarking transposable element annotation methods for creation of a streamlined, comprehensive pipeline. Genome Biology 20, 275.